Les mutations repérées plus vite chez les populations de septoriose
La méthode mise au point à Arvalis permet de détecter rapidement l’apparition de mutations indésirées au sein des populations fongiques de septoriose.
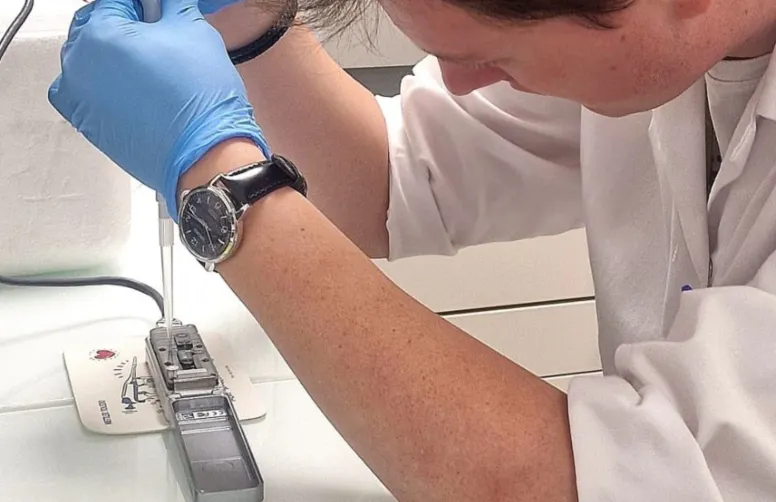
Le projet SeptoMinION 1 s’est achevé en décembre 2025. Il a permis de développer une méthode rapide pour suivre plusieurs gènes d’intérêt chez les populations de Zymoseptoria tritici, champignon responsable de la septoriose du blé. La découverte de mutations génétiques chez les souches de septoriose trouvées au champ peut en effet remettre en cause les stratégies de lutte contre cette maladie.
La méthode moléculaire mise au point à Arvalis s’appuie sur l’outil MinION, développé par Oxford Nanopore Technologies : « un séquenceur polyvalent et abordable dont le laboratoire GenoPaV d’Arvalis s’est équipé », précise Alexandre Leharanger, ingénieur au laboratoire chargé de la mise au point de la méthode.
Sept gènes de Z. tritici sous haute surveillance
Les feuilles de blés contaminées par des souches de Z. tritici sont prélevées au champ puis traitées au laboratoire avant séquençage. Les séquences sont ensuite analysées bio-informatiquement, afin de repérer d’éventuelles mutations dans les gènes d’avirulence AvrStb6, AvrStb9, AvrStb16q et AvrStb20q (de telles mutations peuvent permettre à la maladie de contourner les résistances variétales), ou dans les gènes CYP51, SDHI et Cytb ciblés par les fongicides, dont les mutations peuvent conférer une résistance aux traitements.
« À plus long terme, il pourra être possible d’adapter finement la lutte contre la septoriose en cours de campagne », conclut Éloïse Méteier, responsable du Laboratoire de Pathologie Végétale d’Arvalis et co-encadrante du projet.
1. Débuté en janvier 2025, le projet SeptoMinION est piloté par Arvalis et financé par le programme Carnot-Plant2PRO.




0 commentaire
Réagissez !
Merci de vous connecter pour commenter cet article.