Variétés de blé tendre : le marquage moléculaire accélère l’évaluation variétale

Au cours des dernières décennies, les méthodes et les outils moléculaires se sont fortement développés pour explorer le génome des plantes et favoriser l’identification de gènes responsables de caractères agronomiques comme l’adaptation au stress hydrique ou la sensibilité à une maladie. Les méthodes de sélection assistée par marqueurs(1) et de sélection génomique(2) contribuent à l’amélioration du profil agronomique et de la qualité des variétés. Le séquençage complet du génome du blé, achevé en 2018 par un consortium international, devrait encore accélérer ce mouvement sur cette espèce.
Le marquage moléculaire et l’expérimentation au champ sont utilisés de manière complémentaire pour apporter une information fiable et rapide aux utilisateurs.
Un gène peut présenter, au niveau de sa séquence, des variations impactant son effet sur le caractère ; ces variations sont appelées des allèles. Afin de repérer facilement la présence d’un allèle chez une plante, il est nécessaire d’identifier un ou plusieurs marqueurs moléculaires diagnostiques de sa présence. Un marqueur est une petite séquence d’ADN spécifique d’une région du génome facilement repérable à l’aide d’outils moléculaires. Pour être diagnostique, c’est-à-dire capable de prédire le comportement au champ des variétés, le marqueur doit être fortement lié à la présence de l’allèle du gène cible, autrement dit être situé sur ce gène ou à proximité immédiate, rendant ainsi les recombinaisons génétiques très peu probables (figure 1).
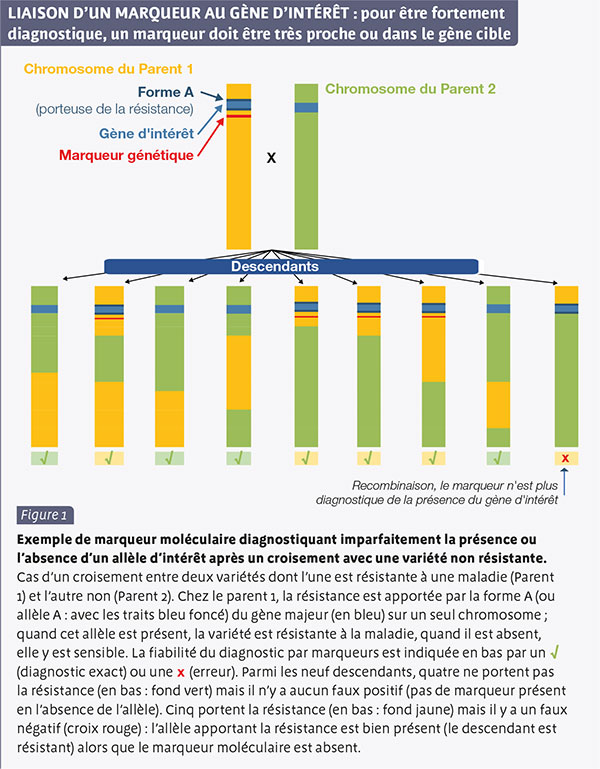
Plus un marqueur est proche de l’allèle d’intérêt, plus les deux seront liés, et plus faibles seront les proportions de faux positifs (par exemple, présence du marqueur de résistance à une maladie, mais variété sensible) et de faux négatifs (absence du marqueur, mais variété résistante). Les erreurs de diagnostic peuvent également être dues à l’introduction par les sélectionneurs d’une nouvelle source de résistance pour laquelle aucun marqueur lié n’est encore disponible.
Pour être jugé efficace pour prédire le comportement agronomique, un marqueur doit être 100 % diagnostique, car il n'y a alors aucun faux positif ni faux négatif possible.

Piétin-verse, mosaïques, cécidomyie orange : des résistances à déterminisme génétique simple
Pour certains caractères d’intérêt, la gouvernance génétique est dite « simple » car elle n’implique qu’un gène majeur. Dans ce cas, la présence d’un marqueur moléculaire lié au gène en question sera prédictive du comportement de la variété pour ce caractère d’intérêt agronomique. C’est le cas des résistances au piétin-verse (gène Pch1), à la mosaïque des céréales (gène Sbm1), à la mosaïque des stries en fuseau (QTL 2D) ou à la cécidomyie orange (gène Sm1). En sélection, ces marqueurs permettent de trier les plantes qui présentent un ou plusieurs de ces caractères d’intérêt.
Arvalis utilise certains de ces marqueurs (piétin-verse, mosaïques) pour délivrer une information plus rapide et plus précise aux producteurs. En effet, si les variétés de blé tendre inscrites au Catalogue français bénéficient d’une description détaillée de leurs caractéristiques agronomiques, ce n’est pas le cas des variétés issues du Catalogue européen, telles que RGT Sacramento, Creek ou encore Winner. Ainsi, lorsqu’une variété d’origine européenne possède un marqueur favorable, une année de test au champ en situation contaminée suffit dans la majorité des cas pour confirmer son caractère résistant. Les variétés qui ne présentent pas de marqueur favorable ne sont pas testées au champ et sont, par conséquent, présumées sensibles. Le marquage moléculaire et l’expérimentation au champ sont ainsi utilisés de manière complémentaire pour apporter une information fiable et rapide aux utilisateurs.
• Le piétin-verse est une maladie provoquée par un champignon qui attaque la base de la tige, fragilisant le plateau de tallage et pouvant entrainer la verse de la plante. Première intervention phytosanitaire, le traitement du piétin-verse a souvent lieu à un stade trop précoce (1-2 nœuds) pour être également efficace sur la septoriose. Un traitement anti-piétin conduit donc souvent à une intervention de plus. Choisir une variété résistante à ce champignon fait baisser l’IFT et les charges. Dans les situations à risque élevé, cette stratégie est d’autant plus importante que le traitement fongicide est, au mieux, efficace à 50 %.
La résistance variétale au piétin-verse est apportée par le gène Pch1 situé sur le chromosome 7D. Bien qu’introduit depuis plus de 40 ans dans les variétés, aucun contournement de ce gène n’a été identifié en France à ce jour. Un marqueur moléculaire a pu être amélioré pour développer un outil performant détectant la présence du gène dans les variétés : ce gène est aujourd'hui présent dans environ 20 % des variétés cultivées en France. Les variétés dont la présence du gène est révélée par marquage sont cotées avec une note de résistance au piétin-verse au moins égale à 5 (soit un bon niveau de résistance) et ne nécessitent pas d’application de fongicide spécifique de ce risque. À l’inverse, les variétés actuellement cultivées en France ne possédant pas ce gène ont une note de résistance inférieure ou égale à 4. Dans les principales variétés cultivées en France, il n’existe à ce jour pas d’autre gène que Pch1 permettant d’atteindre un niveau satisfaisant de résistance au piétin-verse.
• Les symptômes de mosaïques sur blé tendre peuvent être induits par deux virus différents, transmis par un pseudo-champignon du sol, Polymyxa graminis : la mosaïque des céréales et la mosaïque des stries en fuseau. En réduisant le système racinaire dès sa mise en place, les virus affaiblissent fortement les plantes, affectant le rendement et ses composantes. Les parcelles infestées le sont de manière quasi définitive car il n’existe aucun moyen de lutte contre le parasite vecteur, dont la forme de conservation peut attendre plusieurs dizaines d’années dans le sol. La résistance variétale est le seul moyen de lutte.
Deux marqueurs génétiques liés à la résistance à ces deux virus sont actuellement disponibles. Le premier détecte la présence du gène Sbm1 qui confère la résistance à la mosaïque des céréales et qui est présent dans environ 15 % des variétés cultivées en France actuellement. Si d’autres sources de résistance à ce virus existent dans les variétés (Sbm2), aucune ne semble aussi efficace que Sbm1 vis-à-vis des souches de virus présentes sur notre territoire. Sur le large panel de variétés testées avec ce marqueur, il existe un faible pourcentage de faux négatif et de faux positif qui peuvent être expliqués par plusieurs hypothèses : une erreur expérimentale sur le phénotypage ou sur le génotypage, une recombinaison génétique entre le marqueur et l’allèle de résistance, ou la présence d’un autre gène de résistance très efficace sur ce caractère.
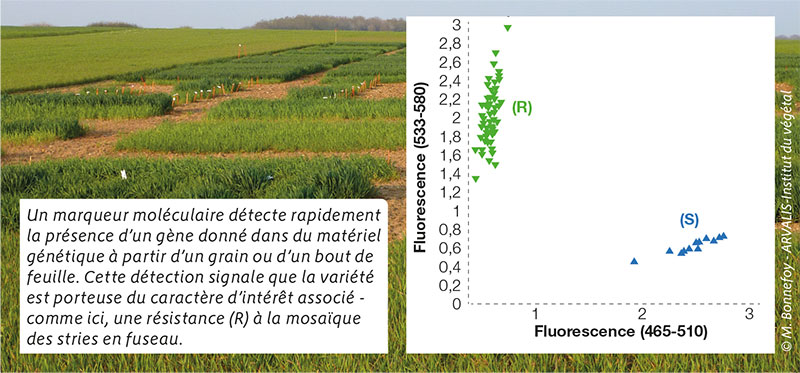
Le marqueur de la résistance à la mosaïque des stries en fuseau est lié à un QTL (une petite zone d’intérêt sur le génome) identifié sur le chromosome 2D du blé en 2018. Le marqueur permet d’identifier 100 % des variétés résistantes et est présent dans environ 85 % des variétés actuellement cultivées. Afin de ne pas faire courir de risque aux producteurs, seules les variétés indemnes aux deux mosaïques sont déclarées résistantes, les autres sont déclarées sensibles au complexe mosaïques.
• La cécidomyie orange, Sitodiplosis mosellana, est une mouche qui occasionne des pertes de rendement via ses larves qui se développent en se nourrissant des grains de blé en formation. Sur blé tendre, la résistance à la cécidomyie orange en France est gouvernée par un gène majeur, Sm1.
Les premières variétés résistantes ont été inscrites au début des années 2000 et aujourd’hui, entre cinq et dix nouvelles variétés résistantes sont inscrites chaque année.
Un marqueur lié au gène de résistance Sm1 a été développé et apparaît comme étant 100 % diagnostique de la résistance observée au champ ; autrement dit, toutes les variétés résistantes possèdent Sm1, et toutes les variétés qui ne le possèdent pas sont sensibles.
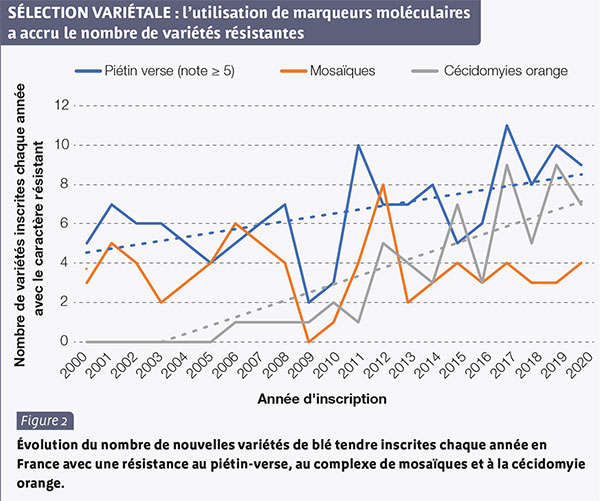
La généralisation de l’usage de ces marqueurs en sélection se traduit par l’augmentation du nombre de variétés résistantes au piétin-verse et à la cécidomyie orange (figure 2).
Quand un caractère d’intérêt dépend de plusieurs gènes
Néanmoins, ces trois cas concrets concernant des marqueurs associés à un unique gène majeur de résistance ne doivent pas masquer la réalité. En effet, la plupart des caractères d’intérêt agronomiques - comme le rendement, la précocité ou la résistance à d’autres maladies (rouilles, septoriose, fusariose) - ont des déterminismes génétiques beaucoup plus complexes. Ils sont « polygéniques », autrement dit contrôlés par plusieurs gènes dont les effets peuvent être soit majeurs, soit mineurs.
Pour tous ces caractères complexes, un ou deux marqueurs génétiques ne suffisent pas à prédire précisément le comportement au champ des variétés. Ces caractères doivent être analysés avec des approches plus complexes de prédiction génomique, basées sur des modèles prenant en compte l’effet de l’ensemble du génome.
Concernant les rouilles, des dizaines de gènes de résistance ont été identifiés, dont un bon nombre sont présents dans les variétés cultivées en France. L’efficacité de ces gènes est en interaction avec les virulences portées par les populations de rouille présentes sur notre territoire, en constante évolution. Mais cette efficacité est aussi liée au stade de développement des plantes : par exemple, certains gènes de résistance sont efficaces dès le stade plantule, alors que d’autres ne sont actifs qu’au stade adulte.
De plus, les niveaux de résistance partielle diffèrent selon les variétés. Ainsi, même si les gènes de résistance majeurs sont contournés, certaines variétés de blé maintiennent de très faibles niveaux de symptômes grâce à leurs gènes mineurs qui peuvent se révéler très efficaces lorsque leurs actions se cumulent.
Prédire la durabilité d’une résistance
À terme, la meilleure connaissance du déterminisme génétique des résistances aidera à mieux estimer leur durabilité. Des stratégies de gestion des gènes de résistance sont à l’étude afin d’augmenter leurs durabilités et/ou de ralentir les développements épidémiques. Elles visent à diversifier ces gènes de résistance à l’échelle du territoire ou de la parcelle.
Ainsi donc, l’identification des gènes de résistance aux bioagresseurs en céréales à paille par marquage a connu d’importantes avancées au cours de cette dernière décennie. Cela a conduit le GEVES, avec Arvalis, l'UFS et INRAE, à déposer deux projets de recherche CASDAR, CAP GENOPHEN et CAP PHENOGEN, afin de structurer l'utilisation du marquage moléculaire des gènes de résistance aux bioagresseurs dès l'inscription, et de proposer des scénarios combinant le phénotypage et le génotypage afin de mieux caractériser les résistances variétales. Dans ce cadre, la section CTPS « Céréales à paille » a décidé d'intégrer le marquage du gène Pch1 dans le processus d'inscription à partir de la campagne 2020-2021.
(1) Sélection assistée par marqueurs moléculaires : elle est utilisée par les sélectionneurs pour repérer rapidement des gènes grâce à un faible nombre de marqueurs moléculaires dans les descendants et ainsi identifier les meilleures lignées à garder ou à croiser entre elles.
(2) Sélection génomique : elle vise à prédire la valeur génétique de candidats à la sélection en s’appuyant sur l’ensemble des marqueurs du génome. Elle est aussi utilisée pour les caractères plus complexes impliquant plusieurs gènes (tel que la résistance au stress hydrique ou azoté).
Le caractère diagnostique des marqueurs peut évoluer dans le tempsAfin de diversifier les sources de résistance, les sélectionneurs utilisent régulièrement dans leurs plans de croisements des variétés dites exotiques, pouvant apporter d’autres gènes de résistance majeure à ces caractères. D’autres marqueurs devront alors être développés pour repérer ces nouvelles variétés dont la résistance repose sur un autre gène. De même il est possible qu’un de ces gènes soit un jour contourné par des souches plus virulentes. La présence du gène ne sera alors plus synonyme de comportement résistant au champ. Ce risque dépend de sa fréquence d’utilisation en culture, de son mode d’action et de la capacité d’adaptation de l’organisme phytopathogène, aussi, la performance des marqueurs doit-elle être continuellement vérifiée. C’est pourquoi le marquage moléculaire ne peut se substituer complètement à l’expérimentation au champ, y compris pour les caractères à déterminisme génétique simple.




0 commentaire
Réagissez !
Merci de vous connecter pour commenter cet article.