Édition génomique des plantes: SpRY, un nouveau ciseau moléculaire plus universel
Développée ces dernières années, l'édition génomique(1) est un procédé visant à modifier une séquence génétique, c’est-à-dire une partie d’un gène. Les outils moléculaires d’édition sont tous basés sur le même principe : la coupure des deux brins de l'ADN par une enzyme spécifique (un complexe de protéines appelées nucléase), qui est suivie d’une réparation nécessaire à la viabilité des cellules.
Selon le mécanisme de réparation à l’œuvre, la réparation de l’ADN(2) peut être parfaite, ce qui ne conduit alors à aucune modification du génome, ou produire une altération de l’ADN : soit la séquence coupée est « perdue » (délétion), soit une séquence différente de l’originale s’insère au site de la coupure (insertion). Dans les deux cas, le gène est modifié (mutation). Le type de mutation reste largement aléatoire.
Lorsqu’on souhaite modifier un gène en un site précis, il faut tout d’abord connaître la séquence génétique visée par la modification et disposer d’une nucléase qui cible cette séquence. À ce jour, il y a trois types de nucléases, d’où trois outils moléculaires différents d'édition génomique : les CRISPR, les ZFN et les TALEN.

Toaster son pain à moindre risque Une bonne nouvelle pour les amateurs inconditionnels de pain grillé ou de baguette bien cuite. Des chercheurs de l’institut Rothamsted (Grande-Bretagne) ont utilisé les ciseaux moléculaires CRISPR-Cas9 pour modifier le génome du blé afin qu’il ait moins de chance de former de l’acrylamide à la cuisson. Ce composé, partie prenante de l’arôme et de la saveur du pain mais suspecté d’être cancérogène, se forme quand l’asparagine contenu dans la farine est portée à haute température : plus le pain est brun ou le toast grillé, plus il contient d’acrylamide et ce, d’autant plus que le grain était riche en asparagine. CRISPR-Cas9 a été utilisé pour « éteindre » le gène TaASN2 qui synthétise l’asparagine. Chez les blés au génome modifié, la concentration en asparagine dans le grain est considérablement réduite par rapport aux variétés classiques : jusqu’à 90 % en moins dans une des lignées modifiées. Les coordinateurs du projet indiquent qu’il faut encore valider ces résultats par des essais au champ avant de délivrer ce matériel génétique aux sélectionneurs. Il faudra 5 à 10 ans avant que des blés à faible teneur en asparagine arrivent sur le marché.
CRISPR-Cas, l’outil de prédilection
Décrit pour la première fois en 2012, le système CRISPR-Cas est, à l’origine, un mécanisme de défense immunitaire des bactéries et des archées contre les infections virales ; il fragmente les brins d'ADN envahisseurs pour les rendre inopérants. CRISPR (clustered regularly interspaced short palindromic repeats) est une séquence présente dans le génome des bactéries qui contient plusieurs motifs répétés entrecoupés de fragments d'ADN étranger (spacers) ; en cas d'infection virale, les spacers sont utilisés afin de produire des ARN CRISPR qui s'associent à leur tour à un ARN transactivateur et permettent de cibler les séquences de virus reconnus par la cellule hôte. Ce complexe d’ARN guidera précisément l’enzyme Cas (associée à CRISPR) jusqu'à l'ADN envahisseur afin de le détruire. On peut ainsi y voir une analogie avec le système immunitaire des mammifères.
Ce complexe d’ARN possède une séquence d'ADN complémentaire(3) de l’ARN CRISPR (dite protospacer), immédiatement suivie d'un motif de quelques nucléotides appelé PAM (protospacer adjacent motif). La présence de ce motif PAM permet au complexe CRISPR-Cas d’identifier rapidement la séquence-cible et de procéder à la coupure pour inactiver l’envahisseur.
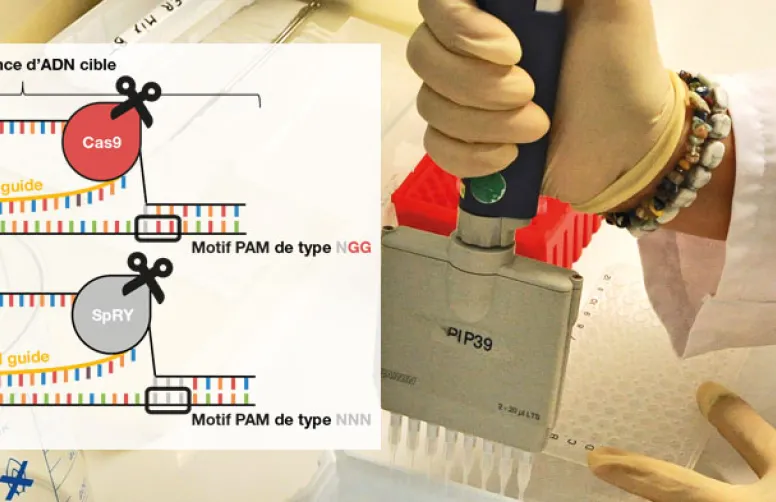
Ce système de défense bactérien a été mis à profit pour en faire un ciseau où l’envahisseur est désormais la séquence à modifier. L’ARN CRISPR et l’ARN transactivateur ont été fusionnés en laboratoire en un « ARN-guide » artificiel comprenant une séquence de reconnaissance de 20 nucléotides, modifiable afin de cibler dans un génome pratiquement n'importe quel gène d'intérêt (en place de l’ADN envahisseur). Cet ARN-guide est associé à la nucléase Cas9, issue de la bactérie Streptococcus pyogenes et optimisée (notée SpCas9) ; il permet au système CRISPR-Cas9 d'être exprimé dans les cellules de tout organisme vivant, et notamment des plantes (schéma).
CRISPR-Cas9 : plus facile à utiliser mais pas « universel »

Comparé aux deux autres types d’outils moléculaires existants, le système CRISPR-Cas9 est beaucoup plus simple et plus facile à adapter à une cible donnée. En effet, la protéine « ciseau » CRISPR demeure toujours la même, seul l'ARN-guide change. Les 20 nucléotides de ce dernier sont choisis et arrangés sur ordinateur selon la cible visée, puis synthétisés par les fournisseurs de réactifs moléculaires. De plus, ce système permet de viser (et donc de modifier) plus d'une cible à la fois si l’on fusionne plusieurs séquences de 20 nucléotides espacées par des répétitions directes comme celles retrouvées dans le locus CRISPR des bactéries.
Toutefois, les actions des enzymes des outils CRISPR-Cas9 sont restreintes aux régions du génome riches en « motifs » PAM, qui précèdent la région cible et qui sont spécifiques à chaque enzyme Cas. Le motif PAM doit être présent sur le brin d’ADN ciblé. La coupure franche sur le brin ciblé survient trois nucléotides en amont du motif PAM.
Le PAM originel, dit « canonique », associé à la protéine Cas9 issue de S. pyrogenes, contient la séquence NGG, où N peut être n’importe quel nucléotide A, T, G ou C. Mais d’autres PAMs peuvent être associés aux protéines Cas9, issus d’autres bactéries telles que Neisseria meningitidis, Treponema denticola ou Streptococcus thermophilus.
Plusieurs tentatives ont été menées pour construire des Cas9 capables de reconnaître d’autres PAMs, afin d’améliorer la capacité d’éditer n’importe quelle zone du génome avec les ciseaux CRISPR-Cas9. Par exemple, le Cas9 de la bactérie Francisella novicida, qui reconnaît le PAM « canonique », a été modifié pour reconnaître une séquence YG (ou Y désigne une pyrimidine), ce qui accroît les cibles possibles de Cas9.
SpRY élargit considérablement les cibles possibles
Récemment, une nouvelle classe d’outils moléculaires dits SpRY(4), également dérivés de la protéine Cas9 de S. pyrogenes, a été développée. Celle-ci semble beaucoup moins spécifique vis-à-vis des motifs PAM. Les SpRY diffèrent des Cas9 par onze nucléotides et sont capables de détecter des motifs de type NAN, NTC, NCT, NCC et NCG. Cela ouvre la possibilité d’éditer des régions jusque-là inaccessibles, comme l’a montré une équipe chinoise notamment sur le riz.
L’outil SpRY est en passe d’être testé sur d’autres espèces végétales afin d’évaluer sa puissance et sa précision. Ces nouvelles études préciseront en particulier le taux d’éditions produites hors cible, qui ont déjà été mises en évidence par l’étude chinoise. En effet, la spécificité des ARN-guide (prérequis à CRISPR) peut conduire à des mutations hors cible, notamment dans les génomes ayant beaucoup de séquences répétées ou dans les génomes polyploïdes comme le blé. Il faudra donc construire des ARN-guides très spécifiques afin d’éviter ces éditions génomiques indésirées et inclure une procédure supplémentaire de criblage pour écarter des schémas de sélection les plantes présentant des mutations hors cible.
(1) Séquence génétique : les gènes sont constitués de quatre sortes de nucléotides : adénine (A), guanine (G), cytosine (C), et thymine (T). Dans les gènes, les nucléotides forment des suites ordonnées, appelées séquences - par exemple, AATGTCA -, aux fonctions spécifiques. Deux séquences sont dites complémentaires si, à chaque nucléotide de l’une, correspond le nucléotide complémentaire dans l’autre : G au lieu de C, C au lieu de G, A au lieu de T, et T au lieu de A.
(2) Séquence d’ARN : (acide ribonucléique). C’est un enchainement dit « simple brin » de quatre nucléotides possibles : A, G, C et U (uracile).
(3)Séquence d’ADN : (acide désoxyribonucléique) « Double brin », elle est formée de deux enchainements des nucléotides A, G, C et T. Les deux brins sont appariés, l’un des brins étant formé des nucléotides complémentaires de l’autre brin (G au lieu de C, C au lieu de G, A au lieu de T, et T au lieu de A).
(4) D’après les articles de L. Hille et B. Kelinstiver, « Plant genome editing branches out » et de Qiurong Ren et al., « PAM-less plant genome editing using a CRISPR–SpRY toolbox », parus en janvier 2021 dans Nature.
Paloma Cabeza-Orcel - p.cabeza@perspectives-agricoles.com
En collaboration avec Matthieu Bogard & Delphine Hourcade (ARVALIS)



0 commentaire
Réagissez !
Merci de vous connecter pour commenter cet article.